環境にはDNAが溢れている
特集 環境DNA 生態系に描かれた生き物たちの航跡をたどって
【環境問題基礎知識】
安藤 温子
環境DNAとは
細胞の中に存在するDNA(デオキシリボ核酸)はアデニン(A)、グアニン(G)、シトシン(C)、チミン(T)と呼ばれる4種類の塩基とリン酸から構成されており、生物を形作るあらゆる情報を含んでいます。DNAは生物の本体だけでなく、そこから放出された断片にも含まれています。例えば動物の場合、排泄物や魚の鱗、鳥の羽、哺乳類の毛や皮膚などが当てはまります。糞には動物が食べたものが含まれますが、その表面には糞を排泄した動物自身の腸内組織などが付着しています。これらの動物の体の断片は、動物たちが日々餌を食べ動き回る中で生息地の環境にDNAを放出し続けています。つまりは土、水、空気など、生物が存在する環境のどこにでもDNAは存在するのです。環境にはDNAが溢れています。このように、生物の体から放出され、環境中に存在するDNAは「環境DNA」と呼ばれています。
DNAにおける塩基の並び順(塩基配列)は、生物によって異なっています。このため、ある場所の土や水などに含まれる環境DNAの塩基配列を解読することで、その場所にどの生物がいたのかを把握することができます。環境DNAは、ある生物がある場所に存在する痕跡、とも言えるでしょう。近年、生物多様性モニタリングの新しい手法として、環境DNA分析が注目されています。環境DNAを用いる利点として、1) 環境中に存在するたくさんの種類の生物を一度に検出することができる、2) 生物を長時間観察する必要がないため、労力が軽減される、3) 捕獲など、生物を殺したり傷つけたりすることなく、その生息状況を把握できる、4) 対象とする生物に関する専門的な知識がなくても、DNAの配列情報をもとに誰でも生物の識別ができる、といった点が挙げられます。それでは、環境DNAを用いた生物調査の方法について、水から魚類を検出する場合を例に紹介します。
サンプリングとDNA抽出
調査地である湖や河川に到着したら、早速採水を行います。採水の方法は調査地によって異なりますが、例えば橋の上から川の水を採取する場合、内側にビニール袋を被せたバケツを川に投げて水を汲み、引き上げた後に専用のボトルに1リットルの水を移します(図1)。水サンプル同士が混ざり合うことを防ぐために、バケツに取り付けたビニール袋を調査地点ごとに交換する必要があります。水中の生物断片に含まれるDNAは時間と共に分解が進むため、出来るだけ手早く作業を行い、採水から1日以内に次の作業に進みます。

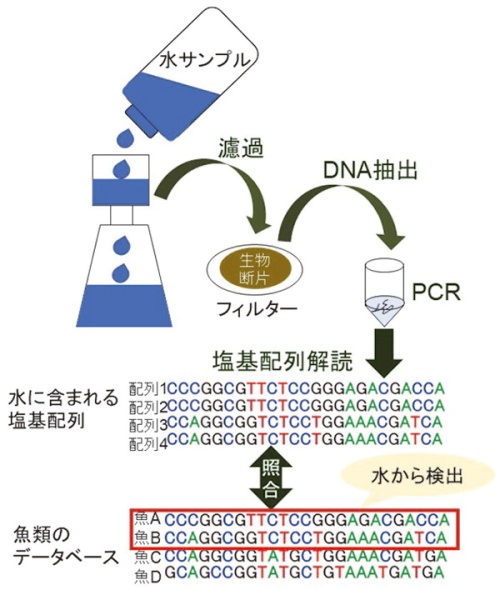
DNAメタバーコーディングによる魚類の検出
抽出したDNAに対して、魚類全般が共通して持つ塩基配列を検出するためのDNA配列(プライマー)で挟まれた領域を、ポリメラーゼ連鎖反応(PCR)という方法によって増幅します。PCRは、DNAが複製する仕組みを応用して、特定のDNA領域だけを増幅させる技術です。DNAはAとT、CとGが相補的に結合して二本鎖を構成していますが、熱を加えることで二本鎖が解離して一本鎖になります。それぞれの一本鎖にプライマーが付着し、DNAポリメラーゼと呼ばれる酵素が働くことで、プライマーを起点とした相補的な配列が構成されます。これを繰り返すことにより、対象とするDNA領域を鼠算式に増幅させることができます。検出したい生物に合わせて様々なプライマーが用いられており、特定の種のみを増幅させるものもあります。今回は、水サンプルに含まれる魚類全般を検出するため、魚類が共通して持つDNA配列をプライマーとして用いています。このように、多くの種のDNAを増幅されることのできるプライマーは「ユニバーサルプライマー」と呼ばれています。増幅が行われたサンプルは、複数の魚類に由来するDNAが混ざり合って存在していますが、次世代シーケンサーという機械を用いて、サンプルに含まれる塩基配列を同時並行的に大量に解読することで、サンプルに含まれる生物を網羅的に検出します。次世代シーケンサーから得られた塩基配列それぞれについて、あらゆる生物のDNA塩基配列が登録されているデータベースと照合することにより、塩基配列が由来する生物を特定します。これらの工程は、DNA塩基配列をバーコードのように用いて、複数の生物の種類を同時に特定することから、「DNAメタバーコーディング」と呼ばれています。DNAメタバーコーディングにより、サンプルに含まれる魚類の種構成を明らかにすることができます。多くのサンプルを分析することにより、魚類の構成の地域的な違い、季節や年による変動を把握することが可能になるのです。
環境DNA分析の課題
水を汲むだけでそこに生息する生物を把握できる、夢の手法のように思われる環境DNA分析ですが、様々な課題も存在します。例えば、サンプリングから実験処理までの工程で、魚類由来のDNAが何らかの理由でサンプルに混入した場合、混入DNAの塩基配列も分析結果に含まれてしまいます。判定された種が調査地に現実的に生息しうるかどうか、慎重に検討してその信頼性を判断する必要があります。生物から放出されたDNAは、時間が経つにつれて分解が進みますが、その速度は調査地の環境によって異なると考えられます。もし、非常に早い速度で分解が進む環境の場合、環境DNA分析によって生物の検出が難しくなる可能性もあるので注意が必要です。また、採取するサンプルの量、回数、場所などについても、調査地の環境や研究目的によって適切な条件が異なるため、慎重な検討が求められます。参照するデータベースが網羅的でない場合、サンプルから得られた塩基配列が間違った種に判定される可能性があります。魚類の場合はかなり充実したデータベースが構築されていますが、他の分類群はその限りではありません。データベースは環境DNA分析による生物調査の基盤となる部分であるため、魚類以外のデータベースも充実させていく必要があります。環境DNAから、調査地に生息する生物の量を評価する方法についても、まだ発展途上の段階です。特定の種に限れば、その種のみを増幅させるプライマーを用いて、増幅の状況からサンプルに含まれているDNA量を算出し、調査地における生物量を推定する方法が用いられています。今回紹介したDNAメタバーコーディングについても、各生物について得られたDNA配列の量から、どの程度の定量化が可能であるかという点について、様々な議論が行われています。環境DNA分析が、より信頼性の高い手法として確立され、多様な環境研究に活用されることを期待したいと思います。
(あんどう はるこ、生物・生態系環境研究センター環境ゲノム科学研究推進室 研究員)
執筆者プロフィール

最近は伊豆諸島で鳥の調査をしています。無人島でキャンプをして、狙っていた鳥を捕獲した時には最高の喜びを感じました。フィールドワーク好きです。


